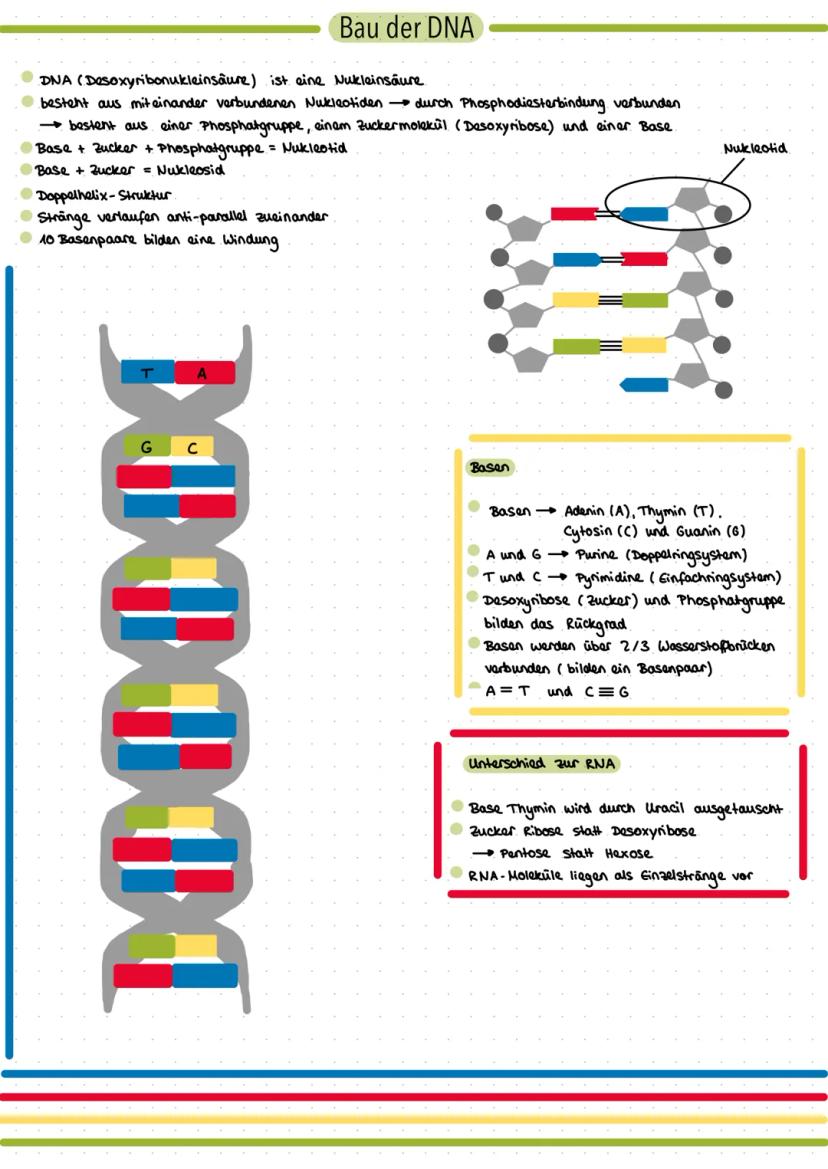
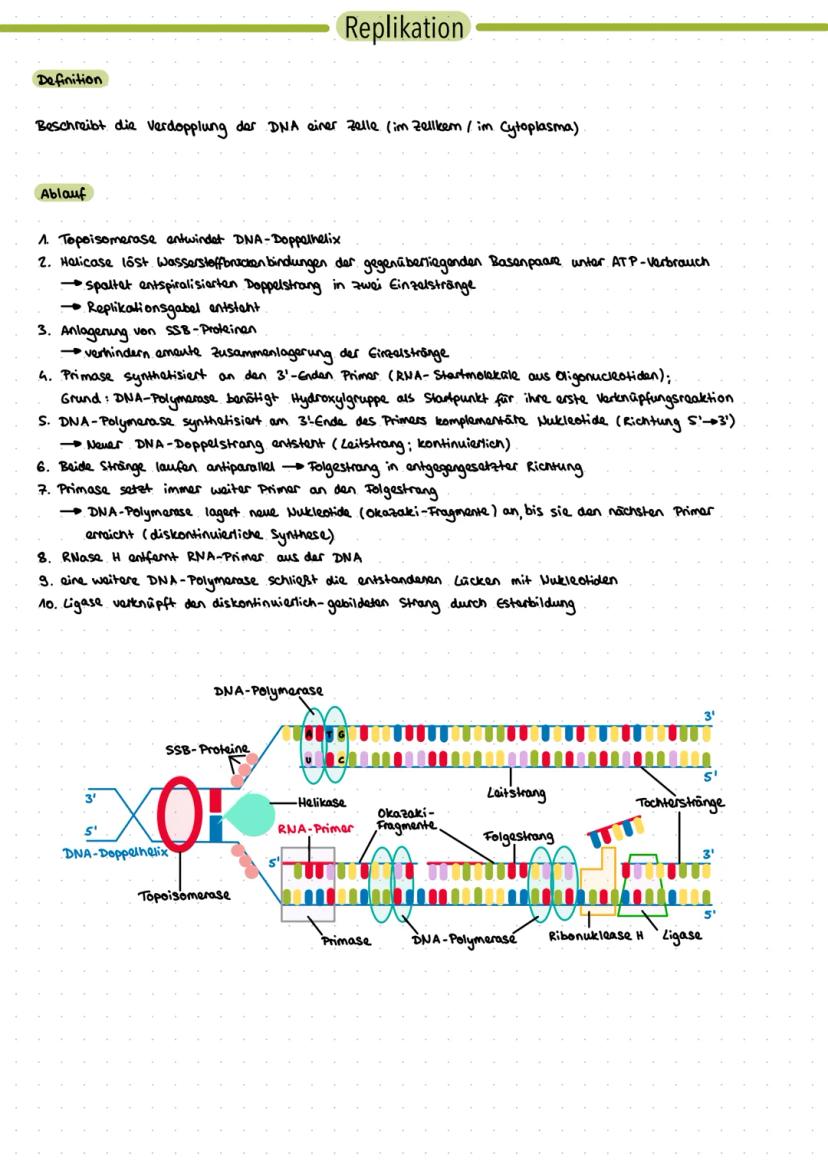
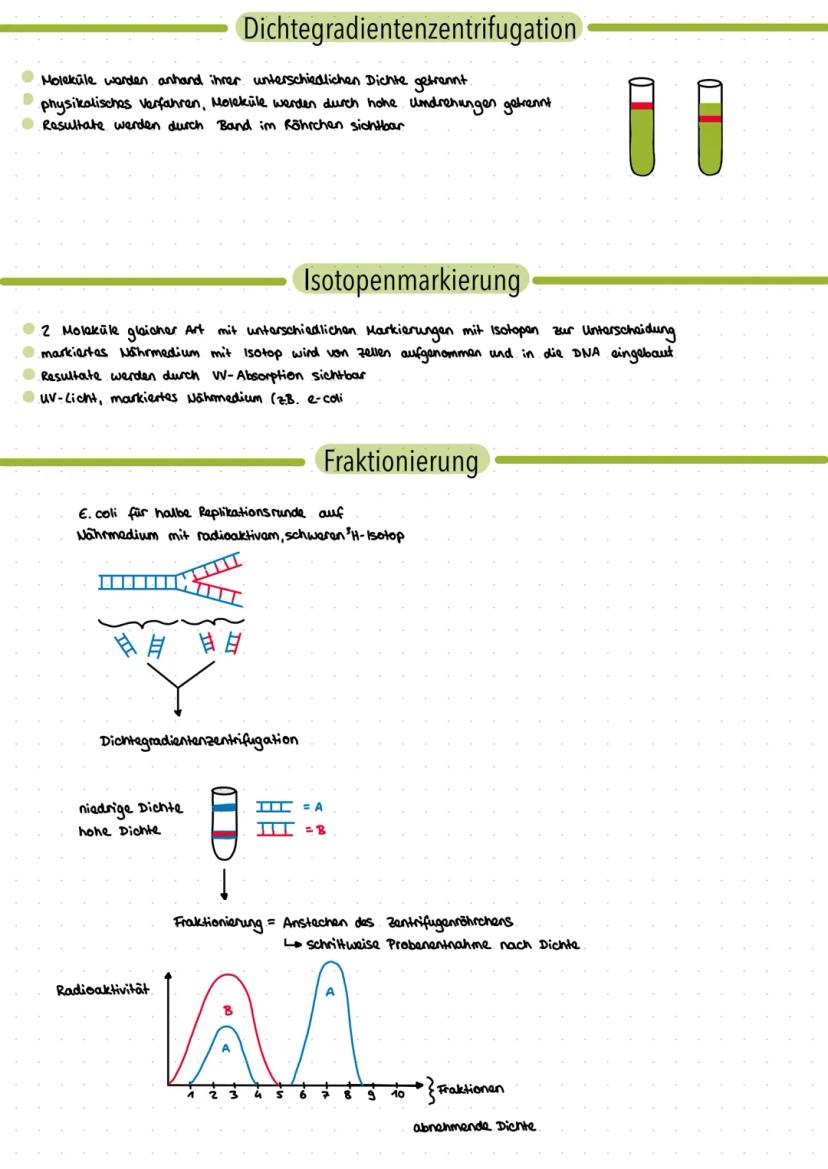
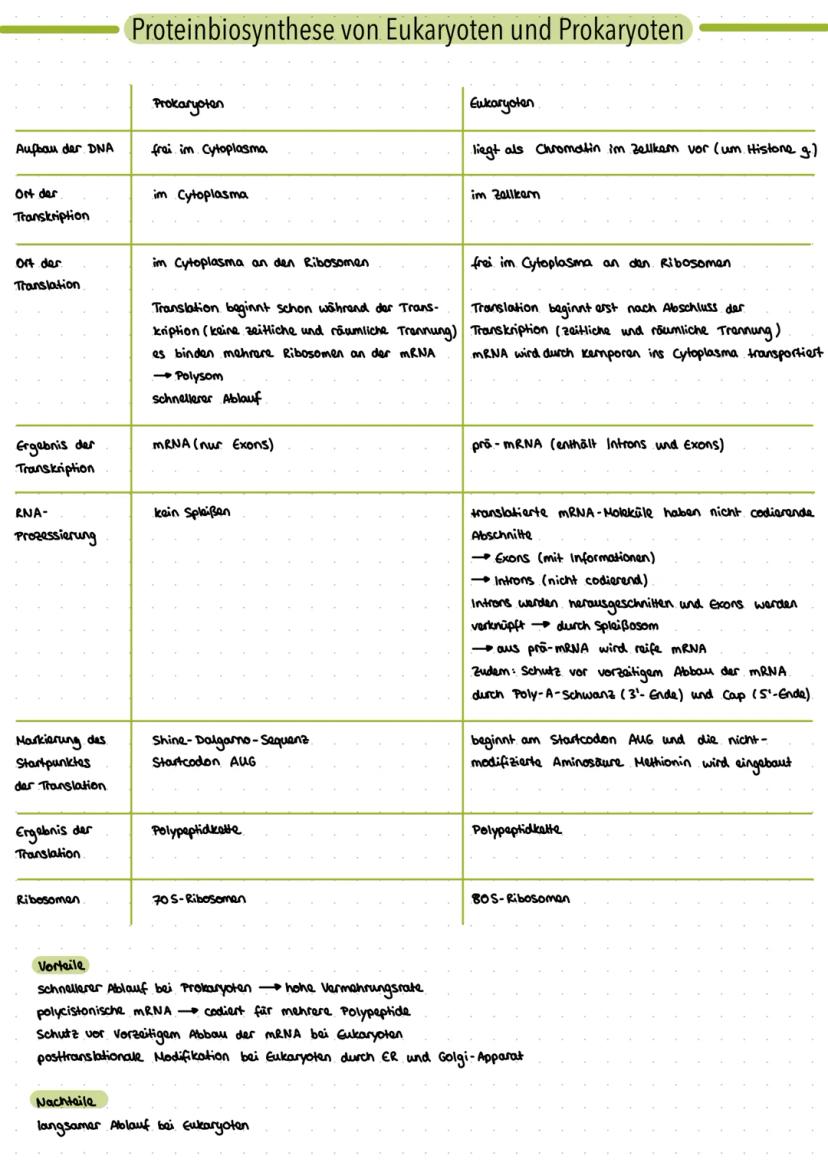
Genetik Lernzettel Q1 fürs Abitur - Bau der DNA - Replikation - Dichtegradientenzentrifugation - Isotopenmarkierung - Fraktionierung - Proteine - Transkription - Translation - Proteinbiosynthese (Prokaryoten/Eukaryoten) - DNA-Veränderung und Reparatur
Melde dich an, um den Inhalt freizuschaltenEs ist kostenlos!
Zugriff auf alle Dokumente
Verbessere deine Noten
Werde Teil der Community
Mit der Anmeldung akzeptierst du die Nutzungsbedingungen und die Datenschutzrichtlinie